東工大ニュース
東工大ニュース
公開日:2015.05.14
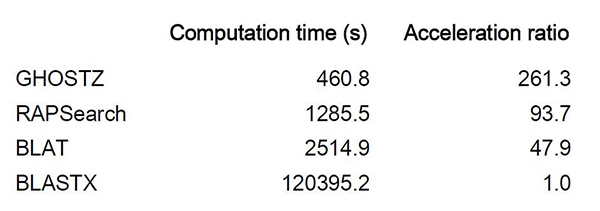
東京工業大学大学院情報理工学研究科計算工学専攻の秋山泰教授らは、ゲノム解析[用語1]で広く使われる配列相同性検索[用語2]を高速に実行する新しいアルゴリズム(問題を解く手順)を開発し、「GHOSTZ」ソフトウェアとして公開した。これは新たに決定した遺伝子等のDNA配列を、すでに決定されている配列のデータベースと比較して類似の配列を見つけ出すソフトウェア。土壌やヒト体内の微生物解析の大規模データで実測したところ、これまで一般的に使われている「BLASTX」ソフトウェア[用語3]に比べて、185~261倍高速に検索できることがわかった。
データベース内の配列をあらかじめ類似配列ごとにクラスタリングして(束ねて)おくことで高感度の比較を保ったままで高速化を実現した。膨大なデータ処理が必要なメタゲノム解析[用語4]も現実的な時間内でこなせる。
配列相同性検索は、ゲノム解析などの基盤となる重要な情報処理である。新しいDNA配列読み取り技術の登場により配列データは日々増加しており、データベースの規模は拡大の一途をたどっている。このため、配列相同性検索に必要な計算負荷は大幅に増加している。
なかでも、土壌・海洋・ヒト体内などの諸環境中に生息する多様な微生物を一網打尽に調査するメタゲノム解析は、得られるデータ量の多さもさることながら、DNA配列が多数の生物種の混合であり、データベース内に近縁の参照配列が存在しない場合が多いことなどから、感度の高い相同性検索を実施する必要があり、計算負荷が特に高くなる。必要な感度を確保できる既存のBLASTXソフトウェアを利用すると、膨大なデータの処理を現実的な時間内では実行できないという点が大きな問題となっていた。
秋山教授らが開発したアルゴリズムは、まず元データのDNA塩基配列を、考え得る何通りかのタンパク質アミノ酸配列に翻訳した上で、既知のアミノ酸配列のデータベースと高感度な比較を行う。このとき、データベース内に出現する部分文字列(既知タンパク質のアミノ酸配列の一部)をあらかじめ類似配列ごとにクラスタリングしておくことで比較処理を高速化した。検索対象となる配列データから、文字列マッチングの核となる短い部分文字列を探索する過程(シード探索)において、初めにデータベース内に作ったクラスターごとの代表配列とだけ比較を行うことにより、全体の比較回数を大幅に減らすことができる。同時に、その後に実施する文字列伸張と呼ばれる計算負荷の大きなステップに持ち込む比較相手の候補を正確に選び出すことにも成功した。
このとき、部分文字列間の距離に関して成立する三角不等式[用語5]に着目したことで、初めは各クラスターの代表配列とだけ比較を行っても、各クラスター内の他のメンバー配列との距離の下限も正確に推定できるために、他の配列との類似性をさらに計算すべきか否かが瞬時に判断できて、見逃しが生じない。これを実現するために必要なデータベース側の前処理も簡単であり、感度を犠牲にせずに、検索速度を向上できた。
この手法をGHOSTZソフトウェアとして効率的に実装し、コンピュータープログラムのソースコードを公開した。土壌細菌や、ヒト口腔内細菌など、メタゲノム解析の複数の実データで測定したところ、GHOSTZは、RAPSearch[用語6]の2.2~2.8倍高速であり、BLASTXの185~261倍高速であった。
GHOSTZは、主にメタゲノム解析において、遺伝子の機能に関する注釈付けや、生物の分類群に関する注釈付けを支援する目的で設計されている。感度の高い相同性検索が必要となるようなメタゲノム研究において、広く用いられることが期待される。さらに、プロテオーム解析[用語7]などの他の研究領域でも利用することが可能である。
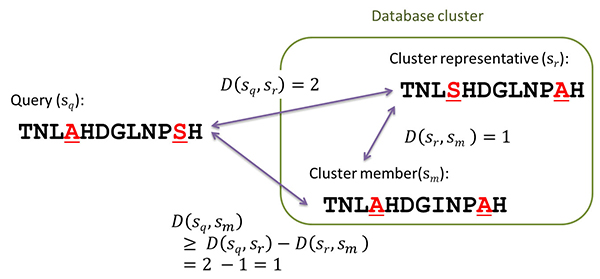
図. 部分文字列間の距離に関する三角不等式を用いた類似度フィルタリング
用語説明
[用語1] ゲノム解析 : ある生物の遺伝情報(DNAの塩基配列)の全体構成と機能を解明する研究。
[用語2] 配列相同性検索 : DNAの塩基配列やタンパク質のアミノ酸配列を調べるときに、すでに配列決定されデータベースに登録されている配列と比較照合して、類似の配列を見つけ出す処理。
[用語3] BLASTXソフトウェア : 配列相同性検索のためのコンピューターソフトウェア。現在、広く使われているソフトウェアである。米国立衛生研究所(NIH)の研究者が製作。
[用語4] メタゲノム解析 : 土壌・海洋・ヒト体内などの環境中に生息する多様な微生物群集について、それぞれの微生物を単離培養せずに、全体としてのゲノム情報を調べる研究。
[用語5] 三角不等式 : A-B間の距離とB-C間の距離の和は、A-C間の距離より大きくなるといった性質を表す数式。本研究の場合は、配列間の編集距離という概念に基づいて式を立てた。
[用語6] RAPSearch : タンパク質のアミノ酸配列に対する配列相同性検索の高速化に特化したソフトウェア。米インディアナ大学の研究者が製作。
[用語7] プロテオーム解析 : ある系(生物種や細胞など)に存在しているタンパク質の全体構成と機能を解明する研究。
論文情報
掲載誌 : |
Bioinformatics 31(8), 1183-1190 (2015) |
論文タイトル : |
Faster sequence homology searches by clustering subsequences |
著者 : |
鈴木脩司、角田将典、石田貴士、秋山泰
|
所属 : |
東京工業大学大学院情報理工学研究科計算工学専攻; 東京工業大学情報生命博士教育院
|
DOI : |
問い合わせ先
大学院情報理工学研究科 計算工学専攻
教授 秋山泰
Email : akiyama@cs.titech.ac.jp
Tel : 03-5734-3645