東工大ニュース
東工大ニュース
公開日:2020.10.14
東京工業大学 生命理工学院 生命理工学系の二階堂雅人准教授、同 総合理工学研究科の張子聡大学院生、国立遺伝学研究所の近藤伸二特任准教授および東京大学 生産技術研究所の甲斐知惠子特任教授らの共同研究グループは、デマレルーセットオオコウモリとエジプトルーセットオオコウモリ(写真)の全ゲノム配列を解読した。
オオコウモリはココウモリの一部から分岐したグループであり、その急速な適応進化については多くの研究者が注目していた。今回の研究では、全ゲノム配列を解読したオオコウモリ2種に加えて、ココウモリを含む22種の哺乳類を対象とした網羅的な比較ゲノム解析を実施し、オオコウモリ独自の進化に関わる遺伝子の探索を行なった。
その結果、オオコウモリのゲノム中において、免疫やタンパク質代謝、そして嗅覚系の遺伝子において適応進化の痕跡[用語1]を検出することに成功した。これらはオオコウモリが持つ高い抗ウイルス免疫力や、果実食という特殊な食性の獲得と深く関連していると考えられる。本結果は、オオコウモリの適応進化について重要な知見を与えるだけでなく、人獣共通感染症ウイルスの感染・発症メカニズムに関わる医学研究への足がかりになると期待される。
本研究の成果は9月23日に『DNA Research』に公開された。

写真. 今回全ゲノム配列を解読したルーセットオオコウモリ
左:デマレルーセットオオコウモリ(Rousettus leschenaultia)
右:エジプトルーセットオオコウモリ(Rousettus aegyptiacus)
写真提供:長谷川政美 統計数理研究所名誉教授
コウモリは完全な飛翔能力を獲得した唯一の哺乳類であり、1,400種を超える莫大な種の多様性を誇っている。そしてコウモリが含まれる「翼手目」は、形態学的な研究によってさらにココウモリ(小翼手亜目)とオオコウモリ(大翼手亜目)の2つに分類されてきた。
ココウモリは最小のもので翼長10 cmほどの小型のグループで、超音波を用いた反響定位能力[用語2]を駆使しながら障害物や餌を探知し、暗闇を飛行する。それに対してオオコウモリは、最大翼長2 mにも達する大型のグループで、反響定位能力を欠く代わりに発達した視覚や嗅覚を用いて餌の探索をおこなう。そして、多くのココウモリは昆虫食もしくは肉食であるのに対し、オオコウモリは果実食または蜜食(花の蜜)に偏った草食である。
このココウモリとオオコウモリは、その間に見られる形態や生態における大きな相違から、かつてはそれらの起源すら異なり、特に視覚神経系の発達したオオコウモリは霊長類に近縁だとする「空飛ぶ霊長類仮説[用語3]」が提唱されたこともあった。しかしDNA配列を用いた分子系統解析[用語4]ではこの仮説は否定され、現在ではココウモリとオオコウモリは近縁であり、さらにオオコウモリは一部のココウモリのグループから派生したことが示されている(図1)。
つまり、コウモリをココウモリとオオコウモリに大別する形態的な分類体系は正しくなく、オオコウモリはココウモリ様の祖先から急速な適応進化を遂げることで、上に述べた数多くの特徴を獲得してきたグループといえる(図1)。また、オオコウモリはニパウイルス感染症やマールブルグ病といった致死性の高い人獣共通感染症を引き起こすウイルスの自然宿主としても知られる。オオコウモリがこうした感染力や毒性の強いウイルスの自然宿主となりうる理由として、このグループが持つ高い抗ウイルス免疫力が挙げられているが、その遺伝的なメカニズムはまだ明らかにされていない。
一般に多くの生命現象には、多数の遺伝子が複雑に入り組んでいるため、その統合的な理解には、全ゲノム情報の解読と網羅的な比較ゲノム解析が有用である。特に、オオコウモリゲノムの解読はこのグループに特徴的な形態や生態の進化のメカニズム解明のみならず、昨今において我々の脅威となっている人獣共通感染症ウイルスの感染・発症メカニズムの解明にもつながると期待される。
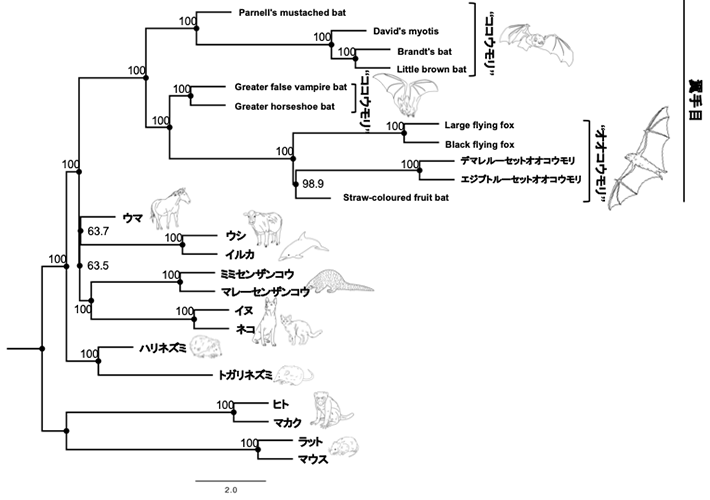
図1. オオコウモリとココウモリと他の哺乳類の系統関係
翼手目(コウモリ)は、大翼手亜目(オオコウモリ)と小翼手亜目(ココウモリ)に分類されていたが、この系統樹が示すようにオオコウモリは一部のココウモリと近縁であり、かつての亜目レベルの分類は正しくないことが分かる。また、空飛ぶ霊長類仮説ではオオコウモリは霊長類(ヒトやサル)と近縁だと予想されたが、この系統樹が示す通り、最近の分子系統解析ではその仮説は支持されない。コウモリ類の詳細な種名は英名のままとした(本研究チームがゲノムを解読した2種は日本語名)。
本研究ではデマレルーセットオオコウモリとエジプトルーセットオオコウモリの2種のゲノム情報を解読した。このオオコウモリ2種に加えて、すでにゲノムの解読が進められていた6種のココウモリおよび3種のオオコウモリを含んだ計22種の哺乳類に関して網羅的なゲノム比較解析することで、オオコウモリの特徴的な進化をもたらした遺伝子群を探索していった。
まず、オオコウモリの化学感覚系遺伝子群(味覚、嗅覚、フェロモン)に着目した。化学感覚を司る遺伝子は多重遺伝子族[用語5]を形成しており、その感覚に対する依存の程度に応じて遺伝子の数が大きく変化することが知られている。各種哺乳類のゲノム中における化学感覚遺伝子の数をそれぞれ比較したところ、特に嗅覚系の遺伝子がオオコウモリのグループで顕著に増加していることが明らかとなった(図2)。
この嗅覚系の遺伝子数の顕著な増加は、オオコウモリが反響定位能力を失ったことによる代替え感覚の進化や、匂いを放つ果実食への適応を反映していると考えられる。
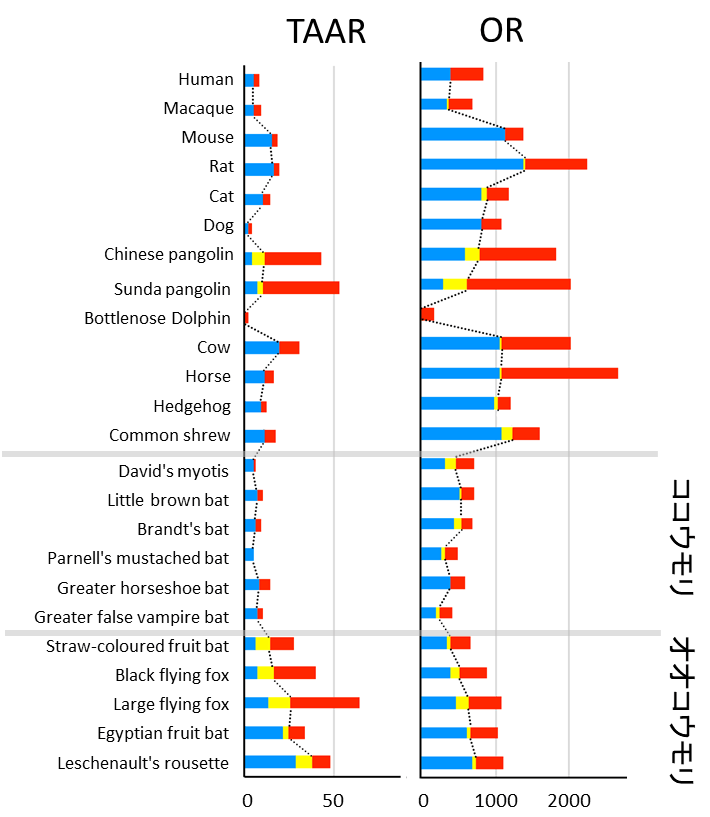
図2. オオコウモリの嗅覚遺伝子の増加
青色と黄色は機能していると予想される遺伝子、赤色は壊れた遺伝子の数(横軸)を示す。ココウモリと比較してオオコウモリは特にOR(olfactory receptor)とTAAR(trace amine-associated receptor)に代表される嗅覚遺伝子が顕著に増加していた。
続いて、オオコウモリの各遺伝子に対して網羅的な選択圧解析を行ったところ、免疫系とタンパク質代謝系の遺伝子群について、適応進化の痕跡が検出された。オオコウモリはニパウイルス感染症やマールブルグ病などに代表される、致死性の高い人獣共通感染症ウイルスの自然宿主となることが確認されている。オオコウモリ自身がこうしたウイルスに感染しても発症しない理由として、オオコウモリが強靭な抗ウイルス免疫力を進化させているためだと予想されてきた。
今回、免疫系遺伝子群において適応進化の痕跡が検出されたことは、この予想によく合致しており、さらに具体的な遺伝子のリストアップにも成功したことになる。今回の研究では宿主ゲノム中でウイルスのように増殖するレトロポゾン[用語6]の活性が、オオコウモリゲノム中では顕著に低下していることもわかった。これはオオコウモリが自身のゲノム中においてウイルスのような可動因子の増殖を防ぐ積極的なメカニズムがある可能性を示唆している。
免疫系以外ではアミノ酸をエネルギー源に変えるタンパク質代謝反応に関わる酵素の遺伝子群において適応進化の痕跡が検出された。これらの酵素の中からタンパク質の立体構造が決定されているものに関して、変異箇所を詳細に調べたところ、その多くがアミノ酸をエネルギーに変換する反応を鈍化させる位置に蓄積していることがわかった(図3)。
このタンパク質代謝を鈍化させる進化の背景には、オオコウモリがココウモリ様の祖先から分岐する過程での肉食から果実食への適応が挙げられる。つまり、果実は極めてアミノ酸に乏しいため、それを主食とするオオコウモリは常にアミノ酸が不足した状態にさらされていると予想される。オオコウモリはこのアミノ酸の不足した状態に適応するために体内のアミノ酸をエネルギーに変換せずに、タンパク質合成に利用するように進化したと考えられる。
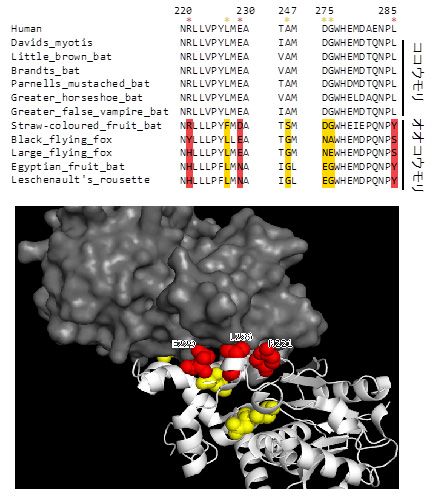
図3. オオコウモリのタンパク質代謝系遺伝子HADHへの変異
黄色はオオコウモリに特有の変異、赤色はその中でも特異性が特に強く示唆される変異。二量体HADHタンパク質のうち、一つ(白)を立体構造で図示し、他方(灰色)を表面構造で図示した。オオコウモリ特有の変異のほとんどは二量体の接合部分に存在、これらの変異が二量体形成を阻害すると予想される。
今回のゲノム比較研究では、オオコウモリの適応進化を嗅覚、免疫、タンパク質代謝系という3つの側面から明らかにすることに成功した。本研究で明らかになった遺伝子については、そのタンパク質の機能に踏み込んだ解析を行うことで、適応進化のメカニズムをより詳細に理解することができると考えられる。
また、2020年に世界的なパンデミックに至った新型コロナウイルスは、人類にとって大きな脅威になっている。今回、オオコウモリゲノム中で検出した免疫系遺伝子群における適応進化の痕跡は、高い抗ウイルス免疫力の獲得と関連していると予想され、人獣共通感染症の感染や発症を防ぐための医学研究への足掛かりにもなると期待される。
研究グループ
本研究は、東京工業大学 生命理工学院 生命理工学系の二階堂雅人准教授、Jiaqi Wu特別研究員、西原秀典助教、同 大学院総合理工学研究科の張子聡大学院生、国立遺伝学研究所の近藤伸二特任准教授、野口英樹特任教授、水口洋平研究員、豊田敦特任教授、藤山秋佐夫特任教授、東京大学 大学院農学生命科学研究科の新村芳人特任准教授、鈴木俊太大学院生(研究当時)、東原和成教授、同 大学院新領域創成科学研究科の鈴木穣教授、千葉大学 未来医療教育研究機構の菅野純夫特任教授、東京大学 生産技術研究所の米田美佐子特任教授および甲斐知惠子特任教授によって進められた。
用語説明
[用語1] 適応進化の痕跡 : 遺伝子に対する塩基置換は、タンパク質のアミノ酸配列を変える変異(非同義置換)と、変えない変異(同義置換)に分けることができる。非同義置換は遺伝子機能に悪影響を与える確率が高く、同義置換は中立と予想される。一般に、機能遺伝子ではその機能を保持するために非同義置換は除去されるように自然選択が働く。ただし、環境の変動などにより、遺伝子が機能を大きく変化させた方が適応的な場合は、非同義置換の中から適応的な変異が選択される。そのため、適応に関わる遺伝子の非同義置換率は通常よりも高くなる。これを適応進化の痕跡として検出することが可能である。
[用語2] 反響定位能力 : 動物が音波を発し、その反響を利用して周囲の環境や獲物の察知、さらには個体間コミュニケーションに用いる方法。哺乳類ではクジラやコウモリが独立にこの能力を獲得しており、反響定位のための独自の器官を発達させている。
[用語3] 空飛ぶ霊長類仮説 : 1986年にペティグリーによって提唱された仮説。オオコウモリと霊長類の間に共通して観察される特殊な視神経の配置パターンは、繰り返し進化するとは考えにくく、これはオオコウモリと霊長類の共通祖先で獲得したものであると予想した。つまり、オオコウモリとココウモリは別の系統であり、飛翔能力も別個に進化したと考えた。その後の分子系統学研究ではこの仮説は支持されていない。
[用語4] 分子系統解析 : DNA配列を比較することで生物の系統関係を推定する方法。分子系統学が確立する以前の生物分類は、主に形態比較に基づいて行われてきたが、研究者の主観が入りやすいという問題点があった。それに対して、あらゆる生物のDNA配列はATGCの4文字で記述されているため、より客観的に系統関係を推定することが可能となった。
[用語5] 多重遺伝子族 : ある1つの遺伝子配列が重複を繰り返すことにより形成された遺伝子ファミリー。重複後の遺伝子は似た機能を持ちながらも個々に進化するため、結果として遺伝子ファミリー内には機能の多様性が生まれる。嗅覚受容体遺伝子ファミリーでは、それぞれの受容体が受容する物質や感度に大きな多様性がある。
[用語6] レトロポゾン : ゲノム中に存在し場所を移動することができるDNA配列の一種。ゲノム中で自分の配列をコピー&ペーストで増殖することができる。ヒトゲノムでは100万コピーを超えるレトロポゾンの配列が存在する。
論文情報
掲載誌 : |
DNA Research |
論文タイトル : |
Comparative genomic analyses illuminate the distinct evolution of megabats within Chiroptera |
著者 : |
Masato Nikaido, Shinji Kondo, Zicong Zhang, Jiaqi Wu, Hidenori Nishihara, Yoshihito Niimura, Shunta Suzuki, Kazushige Touhara, Yutaka Suzuki, Hideki Noguchi, Yohei Minakuchi, Atsushi Toyoda, Asao Fujiyama, Sumio Sugano, Misako Yoneda, Chieko Kai |
DOI : |
お問い合わせ先
東京工業大学 生命理工学院 生命理工学系
准教授 二階堂雅人
E-mail : mnikaido@bio.titech.ac.jp
東京大学生産技術研究所
特任教授 甲斐知惠子
E-mail : ckai@iis.u-tokyo.ac.jp
取材申し込み先
東京工業大学 総務部 広報課
E-mail : media@jim.titech.ac.jp
Tel : 03-5734-2975 / Fax : 03-5734-3661