東工大ニュース
東工大ニュース
公開日:2023.04.11
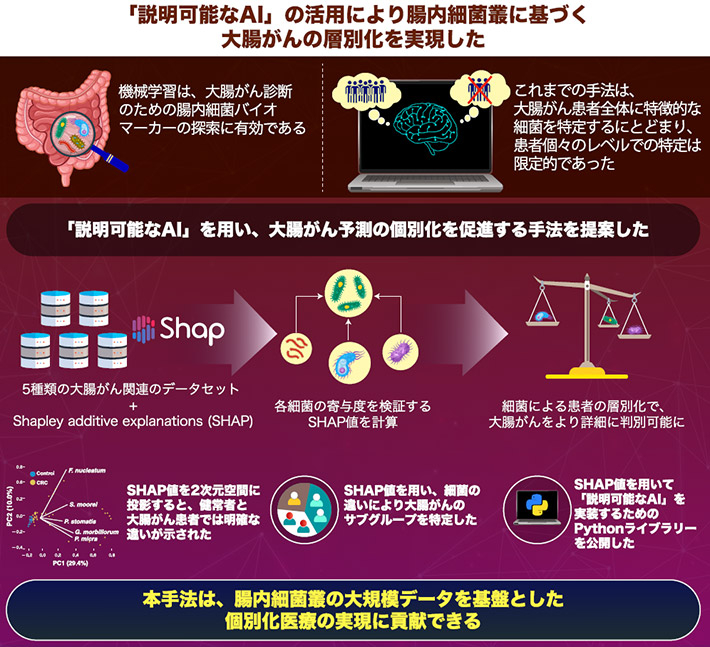
東京工業大学 生命理工学院 生命理工学系の山田拓司准教授、Ryza Rynazal(リザ・リナザル)大学院生(修士課程)(研究当時)、大阪大学 大学院医学系研究科医学専攻 ゲノム生物学教室・がんゲノム情報学の谷内田真一教授らは、「説明可能なAI[用語1]」を活用し、腸内細菌パターンに基づいて大腸がんを詳細に分類する手法を開発した。
現在、腸内細菌データから大腸がんの確率を予測する機械学習モデル(AI)の開発が進んでいる。この場合の機械学習は、腸内環境情報から大腸がんかどうかを判別するモデルと、診断のための細菌バイオマーカーを探索する手法の両方として位置づけられている。
これまでの機械学習モデルでは、大腸がん患者と健常者における腸内細菌群集構造[用語2]の全体的な違いに焦点が当てられることが多く、個々の患者に固有の詳細な腸内細菌群集構造は考慮されていない。そこで研究チームは「説明可能なAI」を利用し、大腸がん患者それぞれに対して「大腸がんらしさ」を表す確率を計算し、その確率に寄与する個人ごとの腸内細菌パターンを見出した。この手法により、大腸がん患者をより詳細なサブグループに層別化することを可能にした。
今回開発した手法は、将来的にはさらにパーソナライズされた大腸がん診断とバイオマーカー同定の実現につながると期待される。
本研究成果は、2月9日付の「Genome Biology」に掲載された。
近年の研究により、腸内細菌群集構造の変化と大腸がんの進行との関連が明らかになっている。大腸がん患者におけるフソバクテリウム・ヌクレアタム(Fusobacterium nucleatum)やパルビモナス・ミクラ(Parvimonas micra)などの特定の細菌の存在量の増加は、大腸がん発症との関連性が報告されている。このような便サンプル由来の細菌情報をバイオマーカーとして利用した大腸がん診断方法の開発も進んでいる。
これまでの大腸がんのバイオマーカー探索や疾患予測では、機械学習モデルがすでに利用されている。機械学習の様々なアルゴリズムの中でも、特にRandom Forestは、予測力や得られた結果の説明の簡易さなどから研究でよく使用されている。機械学習を使ったこれまでの研究報告でも、大腸がんバイオマーカーの最有力候補として、従来の研究と同様にフソバクテリウム・ヌクレアタムが挙げられている。
機械学習を用いたこれまでの解析手法では、こうした大腸がんと関連のある細菌の特定に加えて、細菌群集全体を考慮した疾患確率の計算も可能だ。しかしながら、大腸がん確率に寄与するのはどの細菌かを、それぞれの患者について個別に明らかにすることはできていない。大腸がん患者の中には、フソバクテリウム・ヌクレアタムやパルビモナス・ミクラのような、大腸がん患者一般に特徴的な大腸がん関連細菌は多く存在しないが、別の細菌が健常と大腸がんとの区別を示す場合がある。このように、大腸がん患者の間でも「大腸がんらしさ」に寄与する細菌が異なっているが、これまでの方法では患者ごとという解像度で大腸がんの特徴を捉えることができなかった。
そこで、東京工業大学の山田拓司准教授らの研究チームは、「説明可能なAI」を活用し、腸内環境情報からの大腸がん予測において、重要な情報を層別化し取得する手法を開発した(図1)。これは、ゲーム理論[用語3]の「シャプレー値」に由来するSHAP(Shapley Additive Explanations)と呼ばれるフレームワークを利用したものである。シャプレー値とは、ゲーム理論において、どのようにすればチームを構成するプレイヤー同士で公平に配当を分配できるかを示す値である。同様に、本研究では、大腸がん予測における特定の細菌の影響を示すためにSHAPを使用した。
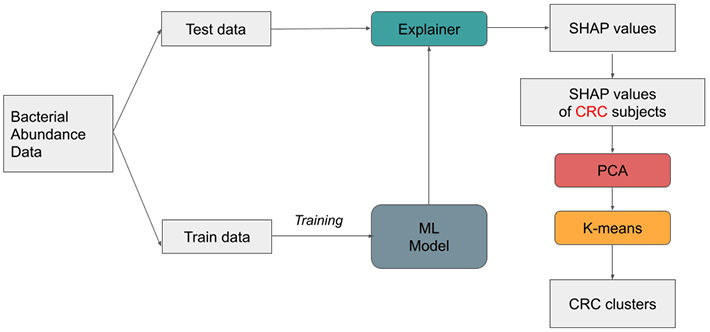
図1. 研究の手法
研究チームは、SHAP値を2次元空間に投影することで、健常者と大腸がん患者を明確に判別できることを発見した。さらに、このSHAP値を用いて大腸がん患者をクラスタリング(層別化)した結果、大腸がん患者が4つのサブグループを形成していることが明らかとなった。この事象を異なる国由来の5つの主要な公開大腸がんマイクロバイオームデータセットを用いて検証し、データセット間で一貫した結果を得ることに成功した(図2)。また、大腸がん確率の値が最も高いサブグループは、大腸がんに関連する細菌も多いことを明らかにした(図3)。
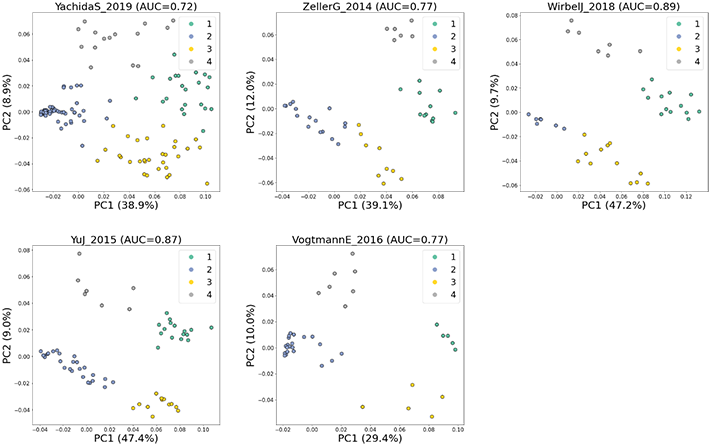
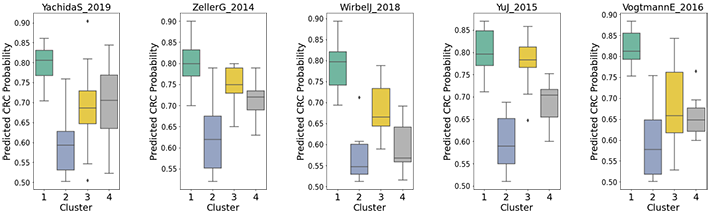
細菌と疾患の関連性の研究において、機械学習アルゴリズムの利用により、疾患の診断に向けた研究が増加している。すなわち、本研究により提案されたこの新しい解析手法は、より層別化されたマイクロバイオームデータを探索し、潜在的な疾患サブグループとそれに関連する潜在的バイオマーカーを見つけ出すのに非常に有益である。
この新しい解析手法は、今後のさらなる研究を通して、よりパーソナライズされた大腸がん診断の実現に貢献するものである。また大腸がんだけでなく、将来は潰瘍性大腸炎や糖尿病、肝臓病など、腸内細菌が関連する他の疾患にも同様に対応できると期待される。
研究の概要
用語説明
[用語1] 説明可能なAI : AI(機械学習やDeep Learningなど)によって判別された事例に対して、その根拠となる特徴量を示すことができる技術。
[用語2] 腸内細菌群集構造 : 多種多様な細菌によって構成されたヒトなどの腸内環境。
[用語3] ゲーム理論 : 事象の相互依存関係をもとにして、それぞれがどのような行動をとるのかを数理的に分析する理論、学問体系。
論文情報
掲載誌 : |
Genome Biology |
論文タイトル : |
Leveraging explainable AI for gut microbiome-based colorectal cancer classification |
著者 : |
Ryza Rynazal1, Kota Fujisawa1, Hirotsugu Shiroma1, Felix Salim1, Sayaka Mizutani1, Satoshi Shiba5, Shinichi Yachida5,6, Takuji Yamada1,2,3,4
|
所属 : |
1 東京工業大学 生命理工学院 2 株式会社メタジェン 3 メタジェンセラピューティクス株式会社 4 株式会社digzyme 5 国立研究開発法人国立がん研究センター ゲノム医科学分野 6 大阪大学 大学院医学系研究科医学専攻 ゲノム生物学教室・がんゲノム情報学 |
DOI : |
お問い合わせ先
東京工業大学 生命理工学院 生命理工学系
准教授 山田拓司
Email takuji@bio.titech.ac.jp
Tel 03-5734-3629 / Fax 03-5734-3591
大阪大学大学院医学系研究科 医学専攻 ゲノム生物学教室・がんゲノム情報学
教授 谷内田真一
Email syachida@cgi.med.osaka-u.ac.jp
Tel 06-6879-3360 / Fax 06-6879-3360
取材申し込み先
東京工業大学 総務部 広報課
Email media@jim.titech.ac.jp
Tel 03-5734-2975 / Fax 03-5734-3661
大阪大学大学院医学系研究科 広報室
Email medpr@office.med.osaka-u.ac.jp
Tel 06-6879-3387 / Fax 06-6879-3399